在动物模型中展现良好治疗潜力

论文截图
南开新闻网讯(通讯员 张光顺 记者 吴军辉)记者获悉,南开大学郭宇、张宏恺、饶子和联合研究团队与上海交通大学附属新华医院、中国科学院武汉病毒研究所以及高诚生物医药(杭州)有限公司等单位合作,成功从康复新冠患者体内分离得到能够高效抑制病毒入侵的中和抗体P4A1、P20A2 和 P20A3,通过X射线晶体学提供的高分辨率结构信息,清晰阐释了中和抗体P4A1的药理机制。该抗体在恒河猴新冠疾病模型中展现出良好的治疗效果。
5月11日,介绍该工作的论文“A SARS-CoV-2 neutralizing antibody with exceptional spike binding coverage and optimized therapeutic potentials”发表在国际知名期刊《自然-通讯》(Nature Communications) 上。
研究团队首先以新冠病毒(SARS-CoV-2)的刺突(Spike)蛋白作为诱饵,从新冠康复患者的外周血单核细胞中分离特异性B细胞,通过单细胞测序方法得到与刺突蛋白结合的B细胞所产生的抗体序列,经进一步筛选得到三个不同的高亲和力抗体P4A1、P20A2 和 P20A3,三个抗体均可以高效结合刺突蛋白S1亚基,抑制S1亚基与病毒入侵受体ACE2的结合。三个抗体在新冠假病毒和活病毒中和试验中均表现出良好的中和活性。
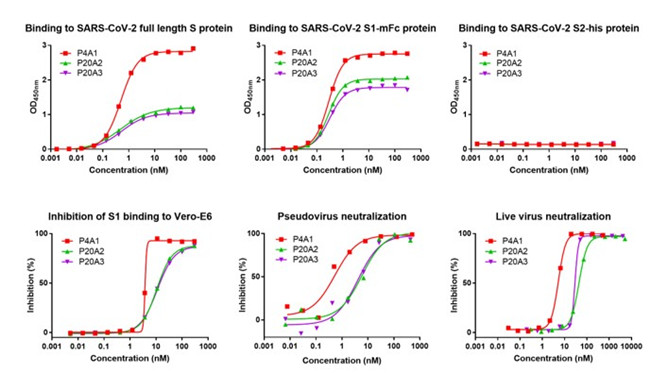
图1 三个中和抗体的体外生物学活性检测
为了探究抗体发挥中和活性的分子机理,该研究团队通过X射线晶体学方法,解析获得了分辨率为2.1 Å的抗体P4A1与新冠病毒刺突蛋白受体结合域(Receptor Binding Domain, RBD)的复合物结构,结果显示P4A1重链上的三个互补决定区(CDR)、轻链上的两个互补决定区,以及3号轻链框架区(LFR3)共同组成了与刺突蛋白结合的相互作用界面,其结合表位覆盖了刺突蛋白上绝大部分细胞受体ACE2的识别表位(占据了17个ACE2识别氨基酸当中的15个),从而有效竞争新冠病毒刺突蛋白RBD与入侵受体ACE2的结合,并进而抑制病毒的入侵过程。
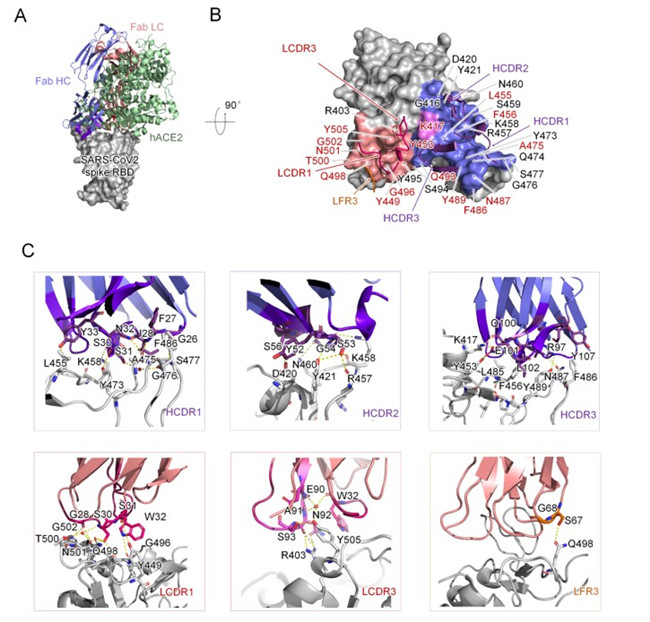
图2 P4A1与SARS-CoV-2 刺突蛋白受体结合域的复合物结构
值得注意的是,为了更好地面向抗体P4A1的临床应用场景需求,该研究团队采用抗体工程改造方法,将其由IgG1型改造为IgG4型,降低多种冠状病毒病中报导的抗体依赖的增强作用的风险并显著延长了抗体的血浆半衰期,在增加安全性的同时为个体提供长期保护。
在此基础上,研究团队与中科院武汉病毒所单超、袁志明团队合作,在新冠病毒感染的恒河猴模型上验证了抗体的治疗效果。结果表明,对照组肺部出现严重支气管肺炎,肺泡腔水肿及大量淋巴细胞浸润,支气管上皮细胞脱落。抗体治疗组则能够有效降低口咽和器官组织的病毒载量并显著减轻支气管肺炎症状,保护正常的肺泡腔结构。
据悉,该改造后的抗体已在美国完成临床I期研究的给药和短期随访,长期随访正在进行。
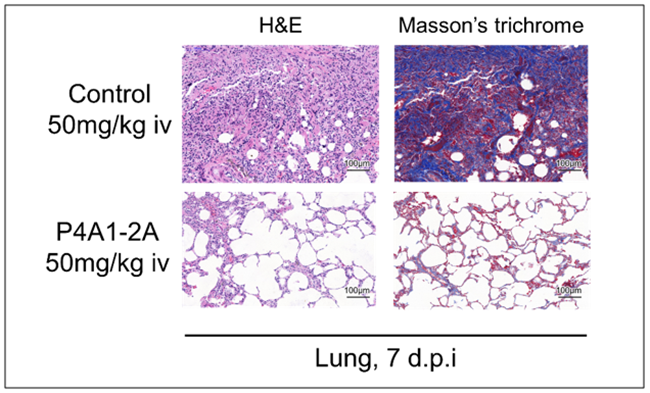
图3 恒河猴模型肺组织病理研究
南开大学郭宇教授为论文第一作者兼通讯作者,上海交通大学医学院附属新华医院黄丽素教授、南开大学博士研究生张光顺、高诚生物医药周禾博士等为论文的共同第一作者;南开大学饶子和院士、上海交通大学医学院附属新华医院孙锟教授、南开大学张宏恺教授、高诚生物医药首席执行官兼首席科学官仲倞(Liang Schweizer)博士等为论文的共同通讯作者。南开大学为论文第一单位。
该研究工作得到了中国科学院武汉病毒研究所、广州实验室、上海同步辐射光源等单位的大力支持,以及国家重点研究计划、国家自然科学基金、天津市自然科学基金重点项目、广州实验室新冠应急攻关项目等项目经费支持。
论文链接:https://www.nature.com/articles/s41467-021-22926-2
|